MÉTODOS
KASP
La tecnología KASP (Kompetitive Alelle Specific PCR) fue desarrollada en principio por KBioScience ahora LGC Genomics (Majeed, 2018; Semagn, 2013). KASP es una plataforma de genotipado uniplex que emplea una forma de PCR de competencia alelo-específica y un sistema de detección basado en fluorescencia para evaluar la variación genética bialélica de SNP (polimorfismos de nucleótido único) o de In/Del (inserciones y/o deleciones) en loci específicos (He, 2014; Semagn, 2013; Robinson, 2011).
La tecnología se basa en la extensión de oligos alelo-específicos y FRET (transmisión de energía de resonancia por fluorescencia) (Semagn, 2013), el ensayo KASP requiere de dos cebadores directos alelo-específicos (un cebador para cada SNP de cada alelo), otro cebador reverso común para ambos alelos y dos oligos, uno marcado con el fluoroforo FAM y otro con HEX, cada oligo es complementario a la secuencia terminal de cada uno de los cebadores de los alelos a evaluar, al iniciar la reacción los oligos fluorescentes están hibridados con desactivadores de fluorescencia. (He, 2014; Robinson, 2011; Majeed, 2018).
En el transcurso de la PCR, el cebador del alelo de interés se une a la hebra molde de ADN y se elonga fijando también la secuencia del extremo terminal donde se hibridará el fluoroforo correspondiente a ese alelo, tras varios ciclos de PCR el fluoroforo pierde su desactivador, se une al cebador directo y emite una señal fluorescente medible. KASP logra discriminar bialelos mediante la unión competitiva de los cebadores directos alelo-específicos, si el genotipo es homocigoto solamente se emitirá una señal fluorescente, si el genotipo es heterocigoto la señal será mixta. (Robinson, 2011).
Animación explicativa de la técnica tomada de la página oficial de LGC Biosearch Technologies
KASP es una de las plataformas de genotipado uniplex más usadas en especies de cultivo debido a su bajo costo y alto rendimiento (Thomson, 2014; Majeed, 2018; Rasheed, 2016; Semagn, 2013) su uso puede mejorar la velocidad y eficiencia de selección en los programas de mejoramiento (Rasheed, 2016). La aplicación de KASP es relevante en ensayos con una densidad de marcadores de baja a media, en análisis de control de calidad, mapeo de QTL en poblaciones biparentales, selección asistida por marcadores y minería de alelos (Semagn, 2013). Los marcadores moleculares convencionales pueden ser convertidos en marcadores KASP, más robustos y de más alto rendimiento (Majeed, 2018). Los ensayos KASP han sido validados para genes de adaptación, rendimiento, calidad y resistencia al estrés biótico y abiótico (Rasheed, 2016).
Protocolo de uso
[Assay Mix]
Allele-1 primer (100 uM): 12 ul
Allele-2 primer (100 uM): 12 ul
Common primer (100 uM): 30 ul
H2O / TrisHCl (10mM, pH8.3): 46 ul
(total volume 100 ul)
[KASP reaction]
DNA (20 – 40 ng/ul): 2 ul
Assay Mix: 0.14 ul
2x Master Mix: 5 ul
H2O: 3 ul
(total volume 10.14 ul)
[PCR condition]
94°C for 15 minutes (Hot-start activation)
10 cycles of
94°C for 20 seconds
61-55°C for 60 seconds (dropping 0.6°C per cycle)
36 cycles of
94°C for 20 seconds
55°C for 60 seconds
Plate reading at 25°C
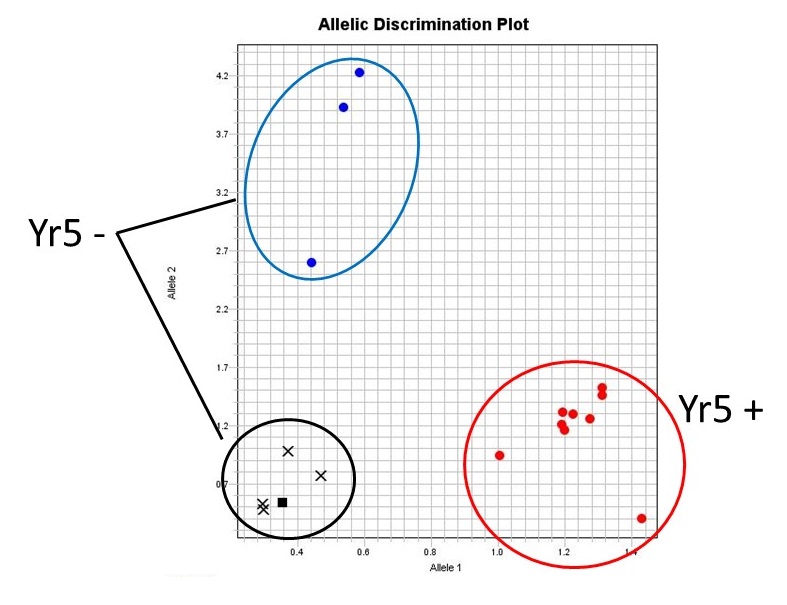
Figura de Trigueros, donde se muestran los resultados de un ensayo KASP.
Referencias
- He, C., Holme, J., & Anthony, J. (2014). SNP genotyping: the KASP assay. En Crop breeding: methods and protocols. D. Fleury and R. Whitford (Eds) pp. 75–86.
- LGC Biosearch Technologies (15 de septiembre de 2021). How does KASP work. https://www.biosearchtech.com/support/education/kasp-genotyping-reagents/how-does-kasp-work
- Majeed, U., Darwish, E., Rehman, S. U., & Zhang, X. (2018). Kompetitive Allele Specific PCR (KASP): A Singleplex Genotyping Platform and Its Application. Journal of Agricultural Science, 11(1), 11. https://doi.org/10.5539/jas.v11n1p11
- Rasheed, A., Wen, W., Gao, F., Zhai, S., Jin, H., Liu, J., Guo, Q., Zhang, Y., Dreisigacker, S., Xia, X., & He, Z. (2016). Development and validation of KASP assays for genes underpinning key economic traits in bread wheat. Theoretical and Applied Genetics, 129(10), 1843–1860. https://doi.org/10.1007/s00122-016-2743-x
- Robinson, P., & Holme, J. (2011). KASP version 4.0 SNP genotyping manual. KBioscience https://www.cerealsdb.uk.net /cerealgenomics/CerealsDB/PDFs/KASP_SNP_Genotyping_Manual.pdf
- Semagn, K., Babu, R., Hearne, S., & Olsen, M. (2013). Single nucleotide polymorphism genotyping using Kompetitive Allele Specific PCR (KASP): overview of the technology and its application in crop improvement. Molecular Breeding, 33(1), 1–14. https://doi.org/10.1007/s11032-013-9917-x
- Thomson, M. J. (2014). High-Throughput SNP Genotyping to Accelerate Crop Improvement. Plant Breeding and Biotechnology, 2(3), 195–212. https://doi.org/10.9787/pbb.2014.2.3.195