GENÓMICA APLICADA
GENÓMICA APLICADA AL MEJORAMIENTO
La llegada de las plataformas de secuenciación de alta escala en los últimos años permitió un avance muy importante en el conocimiento de los genomas de cultivos, entre ellos el trigo cuya primera versión del genoma de referencia secuenciado ha sido presentada en el año 2018 (Appels et al., 2018).
De la mano de estas plataformas, la genómica y la bioinformática, se han acercado cada vez más a los programas de mejoramiento logrando caracterizar profundamente la variabilidad de sus recursos genéticos, promoviendo herramientas de selección genómica y también acelerando notablemente los tiempos necesarios para el descubrimiento de genes de interés agronómico.
El trigo presenta uno de los genomas vegetales más extensos (~17Gb) y más del 80% del mismo, se encuentra compuesto por regiones repetitivas. Estas características hacen que secuenciar un genoma completo de una variedad de trigo sea muy costoso desde el punto de vista económico.
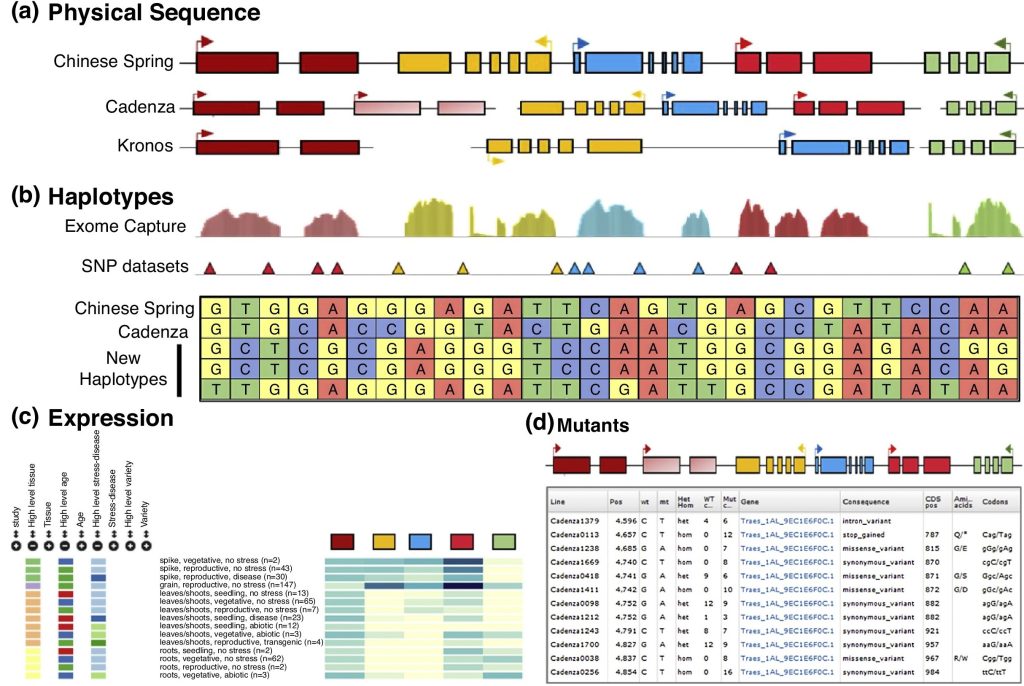
Figura tomada de Uauy (2017).
Para poder reducir los costos y obtener información relevante sobre los genes de las variedades de trigo, se ha desarrollado una técnica denominada captura de exones que permite sustraer del genoma de trigo todas las regiones codificantes para luego realizar una secuenciación de alto caudal, pero solamente considerando dichas regiones (Krasileva et al., 2017).
En esta sección del portal se puede acceder a parte de la variabilidad genética presente en germoplasma latinoamericano y extranjero proveniente de diferentes proyectos de investigación. Esta información permite a los programas de mejoramiento locales conocer la variabilidad presente en la colección, para el 95% de los genes de trigo. Dicha información es muy útil para establecer la presencia o ausencia de alelos de interés para los programas de mejora de una manera rápida y eficiente.
Materiales incluidos
- 31 variedades argentinas analizadas dentro del proyecto de captura y secuenciación de 1.000 exomas de trigo (He et al., 2019).
- Panel Whealbi que consta de 487 genotipos cuyas semillas se encuentran accesibles en el Banco Activo de Germoplasma (BAG) de INTA Marcos Juárez (Pont et al., 2019).
- Padres de la población argentina de mapeo biparental Baguette 19 x BIOINTA 2002 (Pretini et al., 2021)
La información disponible en JBrowse, presenta coordenadas para la búsqueda de genes basados en el genoma de referencia IWGSC Ref Seq v1.0.
Referencias
- Appels, R., Eversole, K., Stein, N., Feuillet, C., Keller, B., Rogers, J., Pozniak, C.J., Choulet, F., Distelfeld, A., Poland, J., Ronen, G., Sharpe, A.G., Barad, O., Baruch, K., Keeble-Gagnère, G., Mascher, M., Ben-Zvi, G., Ambre-Aurore Josselin, A.A., Himmelbach, A., … Uauy, C. (2018). Shifting the limits in wheat research and breeding using a fully annotated reference genome. Science, 361(6403). https://doi.org/10.1126/science.aar7191
- He, F., Pasam, R., Shi, F., Kant, S., Keeble-Gagnere, G., Kay, P., Forrest, K., Fritz, A., Hucl, P., Wiebe, K., Knox, R., Cuthbert, R., Pozniak, C., Akhunova, A., Morrell, P. L., Davies, J. P., Webb, S. R., Spangenberg, G., Hayes, B., & Daetwyler, H. (2019). Exome sequencing highlights the role of wild-relative introgression in shaping the adaptive landscape of the wheat genome. Nature Genetics, 51(5), 896–904. https://doi.org/10.1038/s41588-019-0382-2
- Krasileva, K. V., Vasquez-Gross, H. A., Howell, T., Bailey, P., Paraiso, F., Clissold, L., Simmonds, J., Ramirez-Gonzalez, R.H., Wang, X., Borrill, P., Fosker, C., Ayling, S., Phillips, A.L., Uauy, C., Dubcovsky, J. (2017). Uncovering hidden variation in polyploid wheat. Proceedings of the National Academy of Sciences, 114(6), E913-E921. https://doi.org/10.1073/pnas.1619268114
- Pont, C., Leroy, T., Seidel, M., Tondelli, A., Duchemin, W., Armisen, D., Lang, D., Bustos-Korts, D., Goué, N., Balfourier, F., Molnár-Láng, M., Lage, J., Kilian, B., Özkan, H., Waite, D., Dyer, S., Letellier, T., Alaux, M., Russell, J., & Keller, B. (2019). Tracing the ancestry of modern bread wheats. Nature Genetics, 51(5), 905–911. https://doi.org/10.1038/s41588-019-0393-z
- Pretini, N., Vanzetti, L. S., Terrile, I. I., Donaire, G., & González, F. G. (2021). Mapping QTL for spike fertility and related traits in two doubled haploid wheat (Triticum aestivum L.) populations. BMC Plant Biology, 21(1). https://doi.org/10.1186/s12870-021-03061-y
- Uauy, C. (2017). Wheat genomics comes of age. Current Opinion in Plant Biology, 36, 142–148. https://doi.org/10.1016/j.pbi.2017.01.007